Hydrophobizität, Polarität & Ladungen
Strukturprinzip löslicher globulärer Proteine.
"Hydrophile Schale, hydrophober Kern"
Die Chime-Darstellungen analysieren die Wechselwirkungen eines Globin-Molekül mit der Umgebung. Die "take home message" diese Abschnittes bildet das allgemeine Strukturprinzip löslicher Proteine: Innen hydrophob (Stabilisierung der Tertiärstruktur über van-der-Waals-Wechselwirkungen), außen hydrophil (Bindung von Hydratwasser über Wasserstoffbrücken).
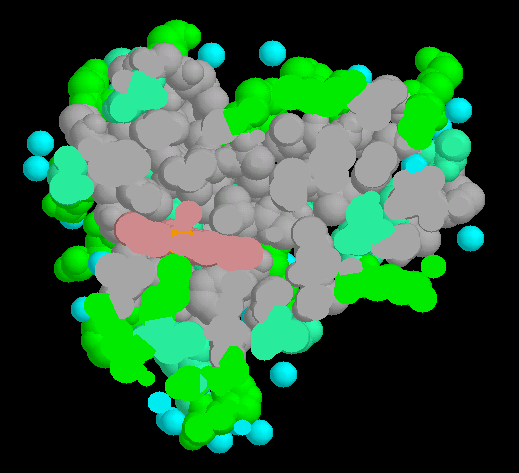
- Die erste Darstellung zeigt die farbkodierte Verteilung hydrophober, polarer und geladener Aminosäuren auf der Globin-Oberfläche sowie die Sauerstoffatome von einem Teil des Hydratwassers. Beim Drehen des Proteins treten hydrophile und hydrophobe Oberflächenabschnitte deutlich hervor. Während die hydrophilen Bereiche mit dem Lösungsmittel Wasserstoffbrücken bilden und das Protein in Lösung halten, stabilisieren die hydrophoben Bereiche über hydrophobe Protein-Protein-Wechselwirkungen zwischen den vier Globinen eines Hämoglobin-Moleküls dessen Quartärstruktur (native Struktur eines aus mehreren Proteinuntereinheiten aufgebauten Proteinkomplexes).
- Der folgende Schnitt macht die Anatomie des Globins - stellvertretend für alle löslichen Proteine - deutlich. Während der Kern durch die Wechselwirkungen hydrophober Seitengruppen stabilisiert wird, ist die dem Medium ausgesetzte Oberfläche mit hydrophilen Resten gespickt.
- Dieses Strukturprinzip wir mithilfe von weiteren Schnittebenen verdeutlicht, die zunächst immer tiefer in das (hydrophobe) Proteininnere vordringen, um sich danach wieder seiner (hydrophilen) Oberfläche nähern (Abb. 8).
Hintergrundinformation: Strukturkodierung und Selbstfaltung
Wie falten sich Proteine?
Die Analyse der Strukturdarstellungen des Globins bietet sich als Ansatzpunkt für weiterführende Fragen zur Proteinstruktur an: Wie finden die linearen Aminosäureketten im lebenden Plasma ihre komplexe dreidimensionale Struktur? Und warum findet dieser Prozess in Zellen mit so hoher Effizienz, im Reagenzglas aber nur mit sehr niedrigen Ausbeuten statt?Vorhersage von Proteinstrukturen
Vom Architekturprinzip der "Packung" einer Polypeptidkette lässt sich leicht der Bogen zur "driving force" ihrer Selbstfaltung schlagen. Der Selbsfaltungsprozess einer Polypeptidkette in ihre native dreidimensionale Struktur wird von ihrer Primärstruktur - also der linearen Abfolge ihrer Aminosäuresequenz - definiert. Dieser Strukturcode ist von Molekularbiologen bis heute noch nicht soweit entschlüsselt worden, dass anhand jeder Sequenz exakte Strukturvorhersagen getroffen werden können (falls das überhaupt möglich ist). In einigen Fällen lassen sich jedoch schon ganz passable Wahrscheinlichkeiten berechnen. All diese Vorhersagen basieren auf einer Bestimmung der thermodynamisch günstigsten Faltung. Das ist zum Beispiel bei einem löslichen Protein (wie vom Globin-Typ) diejenige, die über eine große Anzahl hydrophober Wechselwirkungen im Inneren und hydrophiler Wechselwirkungsmöglichkeiten an der Oberfläche verfügt. Eine gigantische Rechenaufgabe, da im Prinzip die Interaktion eines jeden Aminosäurerestes mit jedem anderen Rest analysiert werden müsste. Die Forscher schränken den Rechenaufwand jedoch erheblich ein, indem zunächst Sekundärstruktur-Wahrscheinlichkeiten analysiert werden. Auch Sequenz-Vergleiche mit Proteinen, deren Struktur bereits durch Röntgenstrukturanalysen eindeutig geklärt ist, erweisen sich als hilfreich: Die Natur verwendet nämlich beim Proteindesign sehr gerne bewährte Proteindomänen (das heißt durch Sekundärstrukturen stabilisierte globuläre Proteinabschnitte, die meist von einem Exon kodiert werden) immer wieder. Aus einem begrenzten Domänen-Repertoire hat die Natur so im Laufe der Evolution eine Vielzahl verschiedener Proteine mit vielfältigen Funktionen "zusammengepuzzelt"."Assisted Self Assembly"
Das auf den bekannten Renaturierungsversuchen von Anfinsen basierende Dogma von der "Selbstfaltung" der Proteine ist seit der Entdeckung der Rolle der "Chaperone" nicht gerade ins Wanken geraten, musste jedoch vom "Self Assembly" zum "Assisted Self Assembly" modifiziert werden. Schnell hatte man erkannt, dass die in vitro beobachteten Selbsfaltungsraten viel zu niedrig sind, um eine Zelle funktionstüchtig zu halten. Zahlreiche Proteine zeigen im Reagenzglas sogar überhaupt keine Neigung, nach einer sanften Denaturierung in ihre native Struktur zurück zu finden. Der Grund dafür ist, dass jede Zelle über ein ganzes Arsenal von Chaperonen verfügt - "molekularen Anstandsdamen" - die mittlerweile auch Einzug in die Schulbuchliteratur gehalten haben. Diese Anstandsdamen (die selbst Proteine sind) erkennen "unordentlich" gefaltete Polypeptidketten, die noch keine stabilen Sekundärstrukturen oder noch keine stabile Tertiärstruktur gefunden haben. Als Symptome solcher unvollständigen oder Fehlfaltungen "fahnden" die Chaperone nach hydrophoben Resten, die an der Oberfläche falsch gefalteter Polypeptidketten exponiert werden. Chaperone entfalten diese unbrauchbaren Gebilde unter Energieverbrauch und verhelfen Ihnen somit zu einer neuen Chance, sich richtig zu falten. Sie "bugsieren" damit den Faltungsweg der Polypeptidketten sicher in die Richtung der thermodynamisch günstigsten Konformation, die in der Regel der nativen Proteinstruktur entspricht.Weiterlesen
- Sichelzellen-Hämoglobin
Molekulare Pathologie der Polymerisation von Sichelzellen-Hämoglobin.